Spezielle DNA-Strukturen erstmals hochpräzise in Lösung vermessen
- Research
- Top News
- Press Releases
- Top-Meldungen
- Forschung
- Medieninformationen

Sie ist eine der bekanntesten Strukturen der Natur: Die Doppelhelix der DNA. Doch das menschliche Erbmaterial gibt es auch in einer viersträngigen Variante, den G-Quadruplexen. Die setzen sich aus Sequenzen zusammen, in denen die Nukleinbase Guanin (G) verstärkt vorkommt, und gleichen einem säulenartigen Stapel. Für die G-Quadruplexe haben Wissenschaftlerinnen und Wissenschaftler in den vergangenen Jahren mehr und mehr biologische Funktionen entdeckt, die auch von medizinischer Relevanz sind.
Um die Rolle der G-Quadruplexe im Körper zu verstehen, muss deren Aufbau und Struktur erforscht werden – eine herausfordernde Aufgabe, da diese speziellen DNA-Formen eine hohe strukturelle Diversität aufweisen. Sind der räumliche Aufbau und die biologische Funktion bekannt, können gezielt Moleküle entwickelt werden, die an diese G-Quadruplexe binden und deren Funktion beeinflussen. Da G-Quadruplexen eine entscheidende Rolle bei der Entstehung verschiedener Krebsarten sowie bei HIV und Malaria zugeschrieben wird, haben DNA-bindende Moleküle das Potenzial, als Wirkstoffe Einsatz in der Medizin zu finden.
Messungen in nie dagewesener Genauigkeit
Bekannt ist zudem, dass G-Quadruplexe auch höhergeordnete Strukturen ausbilden können. Beispielsweise können sie sich paarweise zusammen lagern und sogenannte Dimere bilden. Wenn sich kleine, üblicherweise flache Moleküle in dieses Dimer einlagern, sprechen Forscherinnen und Forscher von „Sandwich-Strukturen“. Diese nur wenige Nanometer kleinen Dimer- und Sandwichstrukturen nachzuweisen und räumlich zu vermessen, ist äußerst schwierig.
In enger Kooperation ist es nun den Dortmunder Gruppen um JProf. Müge Kasanmascheff und Prof. Guido Clever gelungen, eine Methode zu entwickeln, um solche Verbindungen in nie dagewesener Genauigkeit in Lösung vermessen zu können. Zuvor war dies bereits in Kristallstrukturen möglich gewesen, doch die Erforschung in Lösung liegt deutlich näher an der biologischen Realität.
Grundlagen für Suche und Einsatz neuer DNA-bindender Moleküle
Die Teams um Kasanmascheff und Clever bauten dafür gezielt Kupfer-Ionen in chemisch modifizierte G-Quadruplexe ein. Mithilfe der Elektronenspinresonanz-Spektroskopie – JProf. Kasanmascheffs Fachgebiet – konnten die Forscherinnen und Forscher den Abstand zwischen ungepaarten Elektronen der Kupfer-Ionen in den beiden Hälften des G-Quadruplex-Dimers bestimmen. Diese Methode ist in der Erforschung von G-Quadruplex-Paaren und Sandwich-Strukturen zum ersten Mal angewendet worden. In Wasser eigentlich unlösliche kleine Moleküle konnten im Anschluss in die gelösten G-Quadruplex-Dimere eingelagert werden. In der gebildeten Sandwich-Struktur vergrößerte sich daraufhin der Abstand zwischen den Kupfer-Ionen auf charakteristische Weise.
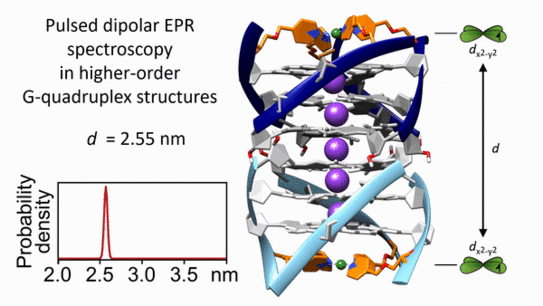
Da die Kupfer-Ionen äußerst fest und starr in den G-Quadruplexen eingebettet sind, konnte das Forscherteam diese Veränderung äußerst präzise messen. Dieses Wissen schafft Grundlagen für Suche und Einsatz neuer DNA-bindender Moleküle zur gezielten Adressierung von G-Quadruplexen.
Die Arbeiten wurden im Rahmen des durch die Deutsche Forschungsgemeinschaft (DFG) geförderten Exzellenzclusters RESOLV durchgeführt und in der Fachzeitschrift Angewandte Chemie veröffentlicht, einem der renommiertesten Journale der Chemie. Die Publikation erhielt die Auszeichnung als „Very Important Paper“, die nur rund fünf Prozent der Veröffentlichungen des Magazins verliehen wird.
Ansprechpartner für Rückfragen: