TU-Chemiker*innen erforschen effiziente Fluoreszenzmarkierung von Proteinen
- Top-Meldungen
- Forschung
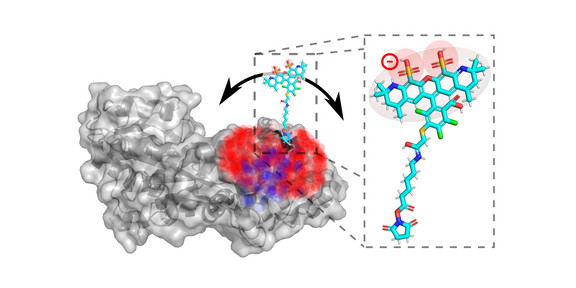
Wissenschaftler*innen untersuchen mithilfe von verschiedenen Verfahren die kleinen Bausteine des Lebens. Um beobachten zu können, wie sich die Struktur von Makromolekülen, wie beispielsweise Proteinen, verändert oder wie sie miteinander interagieren, werden die zu untersuchenden Strukturen an ausgewählten Stellen mit Farbstoffen markiert.
Die Auswahl von geeigneten Markierungsstellen stellt Forschende, aber vor allem Anwendende in der Industrie und Biomedizin, häufig vor Probleme, weiß Prof. Thorben Cordes von der Fakultät für Chemie und Chemische Biologie: „Farbstoffe sind relativ große Moleküle. Sobald sie an Aminosäureresten von Proteinen verankert sind, können sie daher Eigenschaften des Eiweißes beeinflussen sowie zu unerwünschten Wechselwirkungen führen. Das wollen wir vermeiden. Zudem kostet es oft viel Zeit und mehrere Anläufe, geeignete Stellen zu suchen, die sich einerseits für die Fluoreszenzmarkierung eignen, aber andererseits eben nicht die Struktur und Funktion des Proteins beeinflussen.“
Systematische Auswahlmethode
Hier hat das Team um Thorben Cordes und Jan Lipfert von der niederländischen Universität Utrecht angesetzt: Gemeinsam mit Forschenden der Ludwig-Maximilians-Universität München haben die Wissenschaftler*innen aus Dortmund und Utrecht einen methodischen Ansatz entwickelt, um geeignete Markierungsstellen in Proteinen systematisch und effizient zu identifizieren. Dazu haben sie bereits begutachtete Studienergebnisse zu über 100 verschiedenen Proteinen analysiert und Kenndaten von erfolgreich markierten Aminosäureresten zusammengetragen. Cordes erklärt: „Anhand der erstellten Datenbank konnten wir aussagekräftige Parameter identifizieren, mit denen wir durch die Analyse der Proteinstruktur mögliche Markierungsstellen besonders gut bewerten können: die Zugänglichkeit des Restes, die Sekundärstruktur und die konkreten chemischen Eigenschaften der veränderten Aminosäure.“
Um einheitlich auszuwerten, wie gut sich eine Stelle eignet, kombinierte das Team die Parameter in einem „Label Score“, also einem Markierungswert, der beschreibt, ob ein Aminosäurerest zur Klasse der markierungsgeeigneten Aminosäurereste gehört – oder nicht. Diesen quantitativen Ansatz haben die Wissenschaftler*innen anschließend in Experimenten mit verschiedenen Proteinen überprüft und konnten nachweisen: Je höher der berechnete Label Score war, desto wahrscheinlicher eignete sich die Stelle im Vergleich zu anderen dafür, mit Fluorophoren markiert zu werden, ohne das Protein zu beeinflussen.
Frei zugängliches Toolkit
Ihre Erkenntnisse haben Cordes und sein Team in ihrem selbst entwickelten Webinterface „Labelizer“ gebündelt: Es umfasst die Analysedatenbank und berechnet den Label Score anhand der ausgewählten Parameter, basierend auf Strukturen der Protein Data Bank (pdb) oder aus Modellen wie beispielsweise alpha-fold.
Das Webinterface erlaubt die automatisierte Analyse mit interaktiven Elementen wie einer 3D-Visualisierung der Struktur, sodass es auch für Forschende ohne Programmierungserfahrung leicht zugänglich ist. Cordes sagt: „Indem laufend weitere Daten zu erfolgreichen und fehlgeschlagenen Markierungsversuchen ergänzt werden, hoffen wir, langfristig eine zentrale Datenbank aufzubauen, um den Algorithmus immer weiter zu verbessern.“
Zur Publikation in Nature Communication
Ansprechperson für Rückfragen: