Auf die Dynamik kommt es an
- mundo

Um zu verstehen, wie Proteine funktionieren, genügt es nicht, ihre Struktur zu entschlüsseln. Ebenso wichtig ist, wie sie sich bewegen und mit ihrer Umgebung interagieren. Gemeinsam mit internationalen Partnern entwickelt Prof. Rasmus Linser von der Fakultät für Chemie und Chemische Biologie dafür ein Verfahren namens NMR-Spektroskopie weiter, das in der Lage ist, die Bausteine des Lebens in Aktion zu beobachten.
Proteine erfüllen in unserem Körper vielfältige Aufgaben. Sie geben unseren Zellen Struktur und ermöglichen ihnen Bewegungen, transportieren Stoffwechselprodukte, bestimmen unsere Reaktion auf Krankheiten und katalysieren als Enzyme zahlreiche biochemische Reaktionen. Dabei sind sie stets in Bewegung. Denn ihre Aufgaben können sie nur erfüllen, indem sie flexibel ihre Form ändern, um beispielsweise bestimmte Zielstrukturen zu erkennen oder an Reaktionspartner zu binden.
„Wenn wir die Funktion von Proteinen verstehen wollen, ist diese Dynamik essenziell“, sagt Prof. Rasmus Linser von der Fakultät für Chemie und Chemische Biologie. „Proteine in Bewegung zu beobachten, ist allerdings eine große Herausforderung.“ Denn um die dreidimensionale Struktur von Proteinen zu entschlüsseln, sind Bewegungen oft eher hinderlich. Für klassische Methoden der Strukturanalyse werden die zu untersuchenden Proteine deshalb üblicherweise bei extrem tiefen Temperaturen in einem Kristallgitter untersucht. Damit lässt sich zwar der Aufbau der Proteine in hoher Auflösung darstellen, doch ihre Dynamik bleibt verborgen.
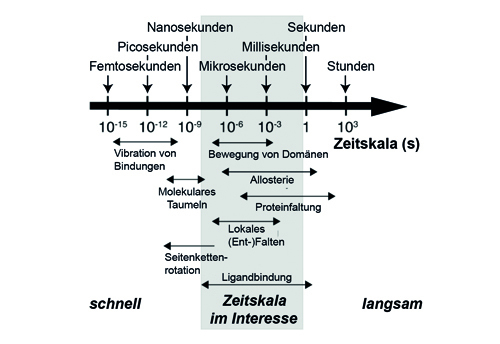
Linser arbeitet daher mit einer anderen Technik: Die sogenannte Kernspinresonanzspektroskopie, kurz NMR-Spektroskopie (nuclear magnetic resonance), ermöglicht, nicht nur die Struktur, sondern auch die Dynamik von Proteinen in einer Probe zu erfassen. „Das physikalische Grundprinzip ist das gleiche wie beim MRT, mit dem in der Medizin Bilder von Knochen und Organen gemacht werden, allerdings mit anderen technischen Facetten“, erklärt Linser. In einem starken Magnetfeld wird eine Probe komplexen Sequenzen elektromagnetischer Pulse ausgesetzt. Aus den entstehenden Spektren lässt sich ableiten, welche Abstände die einzelnen Atomkerne zueinander haben, wie sie in die Proteinstruktur eingebettet sind und wie sie mit Nachbaratomen wechselwirken. Zudem lassen sich Bewegungen auf verschiedenen Zeitskalen erfassen. Von Interesse ist dabei vor allem die Dynamik im Bereich von Millisekunden bis Pikosekunden, in dem die vielfältigen Mechanismen der Proteinfunktionalität häufig ablaufen.
Grenzen ausloten und erweitern
Linsers Forschungsgruppe verfügt dafür über mehrere NMR-Großgeräte, die ein starkes Magnetfeld erzeugen und Proben unter verschiedenen Bedingungen analysieren können. Je nach Einsatzzweck befinden sich die Proteine entweder in Lösung oder werden in Form von Festkörpern untersucht. „Eine der wichtigsten Beschränkungen ist bisher, dass die NMR-Spektroskopie nur für relativ kleine Proteine geeignet ist“, sagt Linser. Denn je komplexer ein Protein ist, desto unübersichtlicher wird das Spektrum. Selbst mit spezialisierten Computerprogrammen ist die Auswertung dann nicht mehr möglich. „In unserer Forschung loten wir die Grenzen des Möglichen aus und versuchen, sie mit innovativen Ansätzen mehr und mehr zu überwinden“, so Linser.
Finanzielle Unterstützung erhält der TU-Professor dabei unter anderem vom Europäischen Forschungsrat (ERC), der Linsers Projekt Anfang 2023 für einen der prestigeträchtigen Consolidator Grants ausgewählt hat und in den kommenden fünf Jahren mit rund zwei Millionen Euro fördert. „Wir kombinieren verschiedene Ansätze, um die Komplexität besser in den Griff zu bekommen“, beschreibt Linser. „Zum einen variieren wir die experimentellen Herangehensweisen – beispielsweise die chemische Natur der Probe, den darauf abgestimmten Einsatz von Hardware und insbesondere die Schemata, nach denen Magnetisierung zwischen Atomkernen hin- und her transferiert und unterschiedlich kodiert wird, die sogenannten Pulssequenzen. Zum anderen setzen wir auch bei der Analyse der Rohdaten an und entwickeln neue Herangehensweisen zur Datenrekonstruktion und -verwertung.“ Dabei ist Linser stets darauf bedacht, die methodischen Fortschritte unmittelbar mit praktischen Anwendungen zu verknüpfen. „Wir kooperieren dazu mit verschiedenen Forschungsgruppen, sowohl innerhalb der TU Dortmund als auch weltweit“, erklärt er.
Internationale Zusammenarbeit
Eines der bisher größten Proteine, die Linsers Team im Rahmen einer internationalen Kooperation mit Hilfe der NMR-Spektroskopie analysiert hat, ist eine modifizierte Form der Tryptophansynthase. Dieses Protein kommt unter anderem in Bakterien vor und ist sowohl für die medizinische Forschung als auch für die Biotechnologie interessant. „Forschungsteams aus Kalifornien und Wisconsin, mit denen wir kooperieren, haben mit Hilfe von künstlicher Evolution eine Variante des natürlich vorkommenden Proteins entwickelt, die für industrielle Prozesse weit effektiver nutzbar ist“, berichtet Linser. „Die Frage ist allerdings: Warum funktioniert das? Die größten Unterschiede liegen dabei meist nicht in der Struktur, sondern in der Dynamik. Und da werden wir hoffentlich einen Beitrag leisten.“
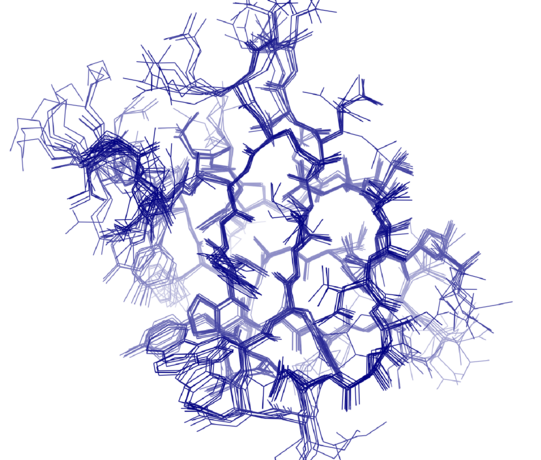
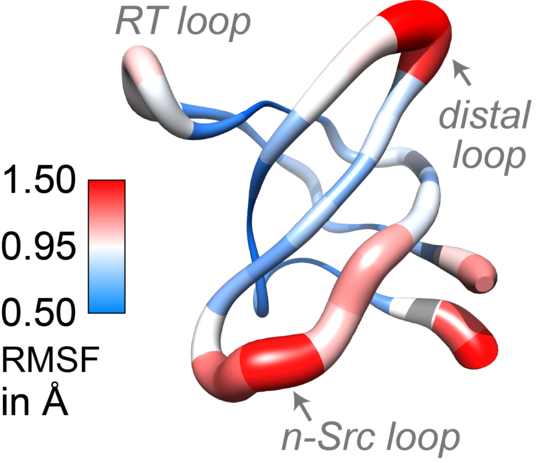
Bisher stieß die NMR-Spektroskopie schon bei Proteinen mit einer Molekülmasse von 40 Kilodalton an ihre Grenzen. Die Tryptophansynthase dagegen besteht aus zwei Untereinheiten mit einer Masse von jeweils 72 Kilodalton – für Linser eine willkommene Herausforderung. Tatsächlich gelang es seinem Team mit neuen experimentellen Ansätzen und einer innovativen Datenverarbeitung, die Grundlagen dafür zu schaffen, die dynamischen Prozesse innerhalb dieser Proteine nun aufzuklären.
Auch im Exzellenzcluster RESOLV, den die TU Dortmund und die Ruhr-Universität Bochum als Sprecherhochschulen koordinieren, arbeitet Linsers Team mit. „Bei RESOLV geht es darum, welche Rolle das Lösungsmittel für verschiedene Vorgänge spielt, beispielsweise in der Katalyse, im elektrochemischen oder im biochemischen Kontext“, erklärt Linser. „Ich interessiere mich vor allem dafür, wie Proteine mit ihm interagieren und es nutzen.“
Einen Fokus hat er in diesem Kontext auf das menschliche Enzym Carboanhydrase gelegt. Die Carboanhydrase katalysiert die Umwandlung von Wasser und Kohlendioxid zu Kohlensäure und andersherum. Damit spielt sie unter anderem eine wichtige Rolle für unsere Atmung sowie für die Säureregulation in Magen und Niere. Entscheidend für die Funktion der Carboanhydrase ist ihr aktives Zentrum, also die Reaktionstasche, in der die Umwandlung stattfindet.

„Mit Hilfe der NMR-Spektroskopie haben wir nachgewiesen, dass das aktive Zentrum eine Konformationsbewegung durchläuft, die auch die ‚erlaubten‘ Plätze der Wassermoleküle in der Tasche beeinflusst“, berichtet Linser. Zuvor war die Carboanhydrase vorwiegend mit Methoden untersucht worden, bei der diese Dynamik verloren geht. Erst die Weiterentwicklung der Möglichkeiten der NMR-Spektroskopie durch Linser und sein Team ermöglichten, das mit rund 30 Kilodalton ebenfalls relativ große Protein mit dieser Technik zu analysieren und dabei Bewegungen auf einer Zeitskala im Mikrosekundenbereich zu erfassen. Erstmals ließ sich so nachweisen, dass das Protein durch seine starke Interaktion mit dem Wasser in der Tasche – trotz der hohen Beweglichkeit der einzelnen Lösungsmittelmoleküle – dem Wasser um das aktive Zentrum herum variierende Eigenschaften aufprägen kann.
Lösungsmittel im Fokus
„Aktuell erforschen wir, wie dynamische Prozesse in unterschiedlichen Elementen der Tasche zusammenspielen. Dabei beziehen wir auch kleine, meist synthetisch hergestellte Moleküle ein, die an das aktive Zentrum binden, sogenannte Liganden“, erzählt Linser. „Das Wasser ist dabei das verbindende Element. Wir wollen genau verstehen, wie die Einheit von Lösungsmittel und Protein die Funktion prägt.“ Auch für die kommende Phase der Exzellenzstrategie ab 2026 hat er schon Ideen. Sofern der Exzellenzcluster RESOLV verlängert wird, könnte er sich gut vorstellen, zu erforschen, wie die interne Dynamik von Liganden, die in ganz unterschiedlicher Funktion an Proteine binden, durch verschiedene Lösungsmittel beeinflusst wird.
„Wir wollen die methodischen Fortschritte unmittelbar mit praktischen Anwendungen verknüpfen. Dazu kooperieren wir mit Forschungsgruppen weltweit.“ Prof. Rasmus Linser
„Solche Liganden können ihr jeweiliges Zielprotein aktivieren, hemmen oder anderweitig seine Funktion modulieren. Oder sie werden als Substrate von dem jeweiligen Protein zu neuen Produkten umgesetzt“, erklärt Linser. „Das Lösungsmittel beeinflusst das Zusammenspiel im biochemischen Kontext dabei wahrscheinlich auf vielfältigere Weise als bisher angenommen: Es könnte gerade größere, komplexere Liganden und Substrate flexibler machen und die Bindung ans Protein erleichtern, oder aber den Prozess erschweren.“ Gerade für die biotechnologische Synthese von größeren Molekülen wären solche Informationen äußerst hilfreich, um die jeweiligen Reaktionen so effektiv wie möglich zu gestalten.
„Bisher lässt sich der Einfluss eines bestimmten Lösungsmittels auf die Molekulardynamik sehr schwer voraussagen“, sagt Linser. „Das Lösungsmittel ist zwar allgegenwärtig, aber seine ortsaufgelöste experimentelle Erfassung ist eine Herausforderung.“ Auch mit der NMR-Spektroskopie wird das Lösungsmittel in der Regel nicht direkt beobachtet – wohl aber sein Einfluss auf die Bewegung der Proteine und ihrer Bindungspartner. „Dieses Feld im Rahmen von RESOLV weiterzuentwickeln, wäre auf jeden Fall sehr reizvoll“, so Linser. „Mit unserem Equipment und unserer Expertise haben wir gute Voraussetzungen, die Forschung auch in diesem Bereich weiterzubringen.“
Text: Elena Bernard
Zur Person:

Prof. Dr. Rasmus Linser ist seit 2018 Professor für Physikalische Chemie an der Fakultät für Chemie und Chemische Biologie. Er studierte Chemie in Göttingen und Madrid und promovierte 2010 am Leibniz-Institut für molekulare Pharmakologie in Berlin. Vier Jahre forschte er abwechselnd an der University of New South Wales in Sydney, Australien, an der Harvard Medical School in Boston, USA, und am Walter and Eliza Hall Institute in Melbourne, Australien. Ab 2014 leitete er eine Emmy-Noether-Nachwuchsgruppe am Max-Planck-Institut für biophysikalische Chemie in Göttingen, von 2016 bis 2018 war er Professor an der Ludwig-Maximilians-Universität München. An der TU Dortmund forscht er an Verbesserungen der NMR-Spektroskopie zur Aufklärung der Struktur, Dynamik und Funktion von Proteinen.
Dies ist ein Beitrag aus der mundo, dem Forschungsmagazin der TU Dortmund.





















